Bạn chưa có tài khoản? Đăng ký ngay

CÔNG NGHỆ TẠO CHỦNG VI SINH VẬT BIỂU HIỆN PROTEIN TÁI TỔ HỢP Ở BACILLUS SUBTILIS
Trung tâm Khoa học và Công nghệ sinh học
- Tối ưu hóa in silico sử dụng các thuật toán, phương pháp Tin-Sinh học để tăng cường sự phiên mã, dịch mã trên chủng biểu hiện.
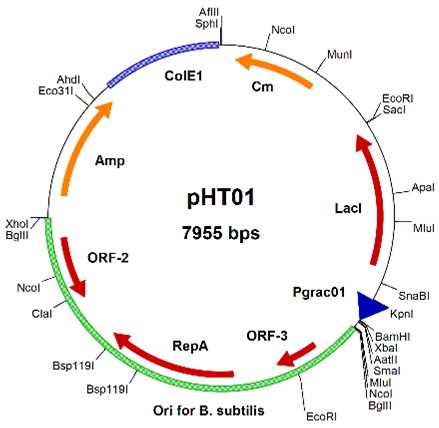
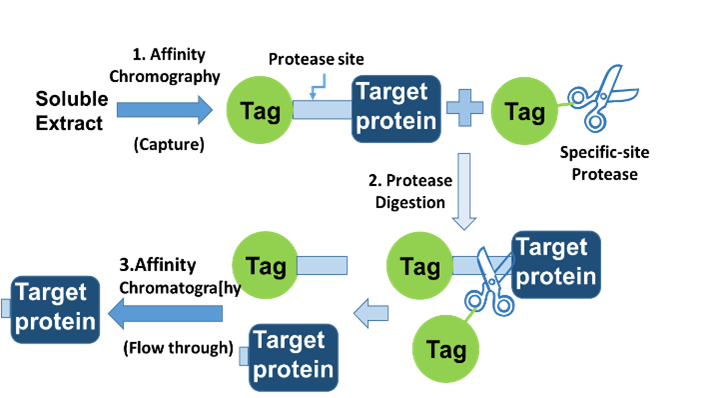
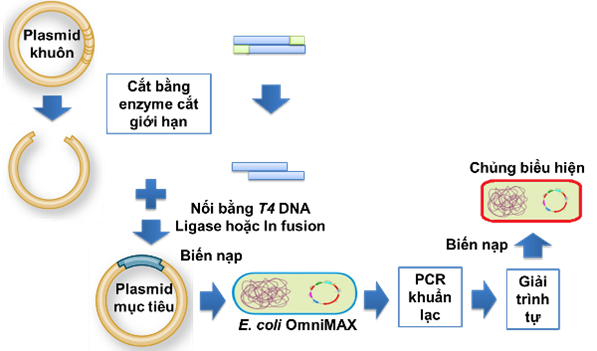
- Gắn gen mục tiêu lên vector (thuộc thư viện vector biểu hiện pHT) có chứa các trình tự promoter (như Pgrac01, Pgrac100, Pgrac212, Pspac, …), Shine-Dalgarno, trình tự đuôi dung hợp nhằm tăng cường sự biểu hiện của protein mục tiêu trên chủng biểu hiện và trình tự tín hiệu để định hướng phương thức biểu hiện (nội bào, tiết, bề mặt tế bào, bề mặt bào tử; liên tục hay cần cảm ứng).
- Các vector mang gen mã hóa protein mục tiêu được sàng lọc để thu nhận bằng phương pháp PCR và giải trình tự.
- Tạo chủng chủ khả nạp (có khả năng tiếp nhận protein mục tiêu). Công nghệ có hai chủng chủ chính là Escherichia coli và Bacillus subtilis-một chủng chủ an toàn không sinh nội độc tố.
- Biến nạp (đưa vector vào chủng mục tiêu). Vector biểu hiện có thể tồn tại ở dạng độc lập như plasmid hoặc sáp nhập vào bộ gen của chủng biểu hiện.
- Sự biểu hiện protein tái tổ hợp của chủng vi sinh vật được xác định bằng SDS-PAGE, Western Blot hay xác định hoạt tính của protein mục tiêu trên môi trường nuôi cấy lỏng hoặc môi trường đĩa thạch.
Thông số kỹ thuật:
- Protein biểu hiện nội bào đạt mức cao nhất lên đến 52 % protein tổng số trên B. subtilis.
- Sự biểu hiện trên bề mặt tế bào, bề mặt bào tử đã được chứng minh.
- Hệ thống có thể giúp protein tiết hiệu quả ở B. subtilis.
Ưu điểm:
- Công nghệ có nhiều lựa chọn để quyết định phương thức biểu hiện protein tái tổ hợp trên chủng vi sinh vật. Điều này thuận lợi để có thể sử dụng cho nhiều mục tiêu như làm vaccine hay sản xuất enzyme quy mô công nghiệp...
- Đặc biệt, các chủng chủ có vector sáp nhập vào genome sẽ không cần các yếu tố áp lực chọn lọc nhằm giữ gen tái tổ hợp không bị đào thải như chủng mang plasmid.